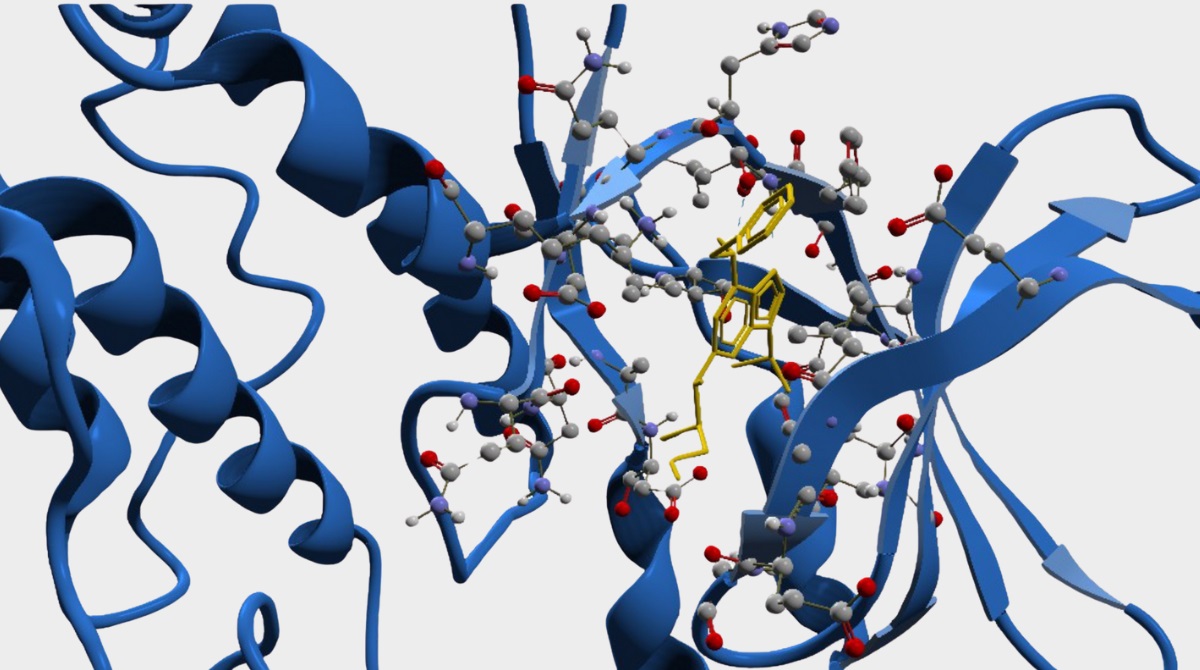
Uma equipe de pesquisadores vinculada aos programas de pós-graduação em Biotecnologia e em Física da UNIFAL-MG utilizou uma ferramenta de IA, chamada SAnDRes, para modelar interações entre a proteína CDK2 (alvo terapêutico no combate ao câncer) e os inibidores, a fim de identificar fármacos em potencial de forma mais rápida e precisa. A pesquisa foi conduzida pelo pesquisador Walter Filgueira de Azevedo Júnior, professor visitante do Programa de Pós-Graduação em Física (PPGF).

Segundo o pesquisador, por meio do SAnDReS foi possível simular o encaixe de inibidores na proteína CDK2 e prever quais moléculas têm chance real de funcionar como fármacos. “Eu resolvi a estrutura dessa proteína com inibidores encaixados nela”, conta Walter Azevedo Júnior. O pesquisador afirma que a informação traz um instante “congelado” com a proteína e o fármaco, tal como fechadura e chave. “Esses dados são tabelados e usados como entrada para criarmos um modelo de aprendizado de máquina”, acrescenta.
A metodologia combina duas tecnologias. Primeiro, a simulação de docking, que é um sistema capaz de testar moléculas que se ligam a essa proteína-alvo. Segundo, o aprendizado de máquina, o qual melhora a previsão ao identificar padrões nos dados. Juntas, essas tecnologias geram uma espécie de pontuação – função escore, que calcula de que forma a molécula se encaixa.
O pesquisador pontua que o uso da abordagem computacional com inteligência artificial permite filtrar as moléculas com maior chance de sucesso. A técnica também contribui para que os pesquisadores concentrem os testes experimentais e clínicos apenas naquelas que já demonstraram bom desempenho nas simulações virtuais. “Todas as fases experimentais e clínicas são otimizadas com o uso da abordagem computacional. Ao invés de testarmos clinicamente milhares de moléculas, testamos somente aquelas que passaram no teste computacional”, argumenta.
Para obter melhores resultados, o pesquisador reforça que os modelos de IA precisam ser treinados especificamente para cada proteína-alvo. “Na nossa experiência e de outros grupos de pesquisa desenvolvendo funções escores ficou claro que aquelas funções ‘treinadas’ para uma proteína específica funcionam melhor que as funções escores universais, que tentam determinar o encaixe para todas as proteínas”, compartilha.
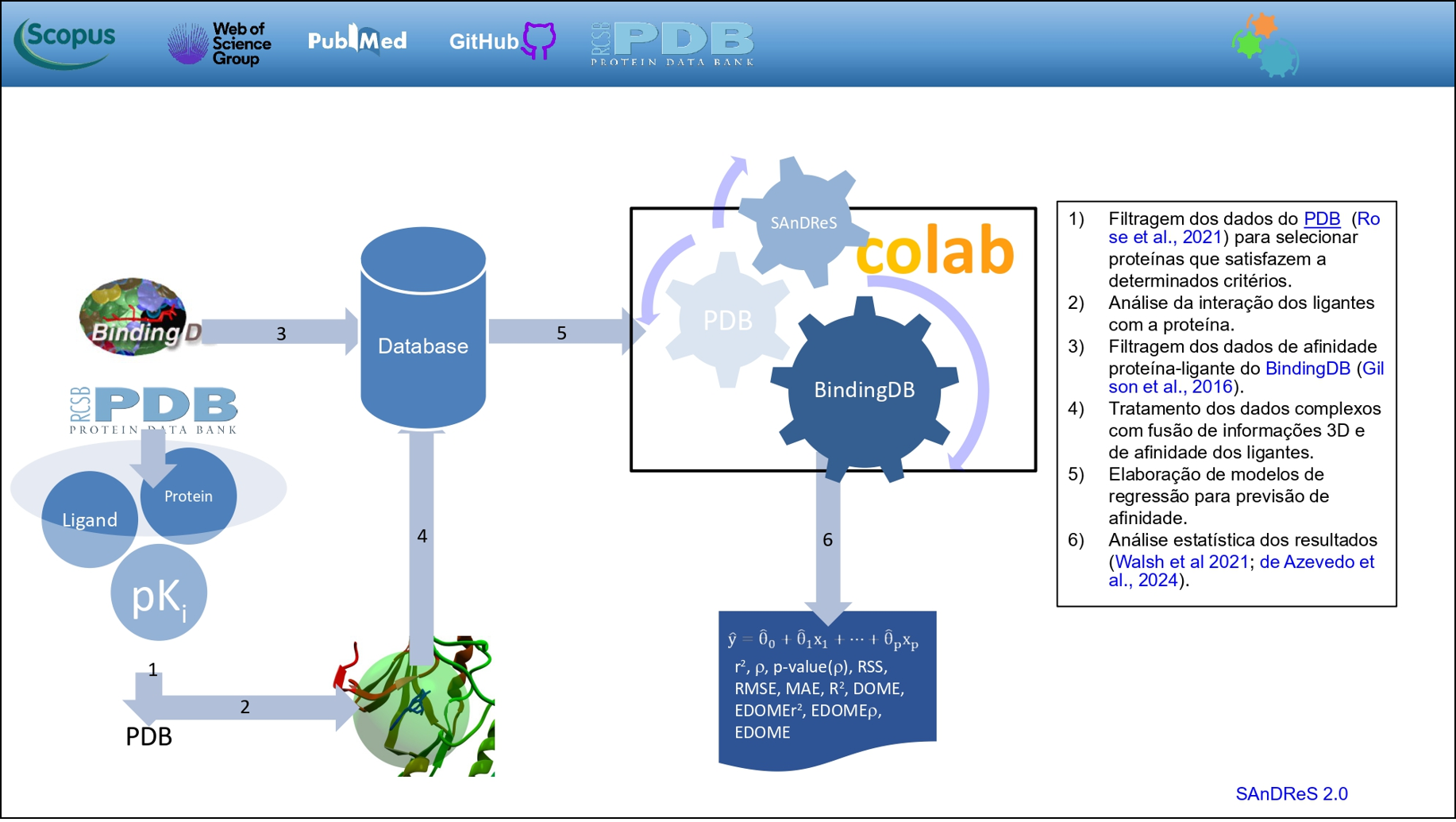
Conforme Walter Azevedo Júnior, o objetivo da pesquisa e do SAnDReS é possibilitar que a comunidade científica tenha acesso a ferramentas como essas, em especial, os grupos voltados a testes de fármacos. O programa SAnDReS é disponibilizado como software livre (open source), o que permite seu uso gratuito em outras pesquisas. Atualmente, seu grupo está ampliando os métodos e as variáveis utilizadas para acrescentar no modelo.
O projeto de pesquisa é financiado pelo Conselho Nacional de Desenvolvimento Científico e Tecnológico (CNPq).
A ferramenta SAnDReS pode ser acessada neste link. Na página Wiki também pode ser encontrada a descrição resumida do programa (em inglês).
Para mais informações sobre as pesquisas e projetos do professor Walter Azevedo Júnior, acesse aqui.

Rafael Martins da Silva Afeto é mestrando em Ciências Ambientais na UNIFAL-MG e bolsista do projeto +Ciência, cuja proposta é fomentar a cultura institucional de divulgação científica e tecnológica. A iniciativa conta com o apoio da FAPEMIG por meio do Programa Comunicação Pública da Ciência e da Tecnologia para desenvolvimento.
*Texto elaborado sob supervisão e orientação de Ana Carolina Araújo